pacman::p_load("data.table",
"tidyverse",
"dplyr", "tidyr",
"ggplot2", "GGally",
"caret",
"gbm") # For gbm
titanic <- fread("../Titanic.csv") # 데이터 불러오기
titanic %>%
as_tibble16 Gradient Boosting
Gradient Boosting의 장점
- 예측 성능이 높다.
- 다양한 손실함수를 최적화할 수 있다.
Gradient Boosting의 단점
- 이상치에 민감하다.
- 과적합이 빠르게 발생할 수 있다.
- 병렬 처리가 지원되지 않아 대용량 데이터셋의 경우 매우 많은 시간이 필요하다.
- 수행시간이 오래 걸린다.
실습 자료 : 1912년 4월 15일 타이타닉호 침몰 당시 탑승객들의 정보를 기록한 데이터셋이며, 총 11개의 변수를 포함하고 있다. 이 자료에서 Target은
Survived이다.
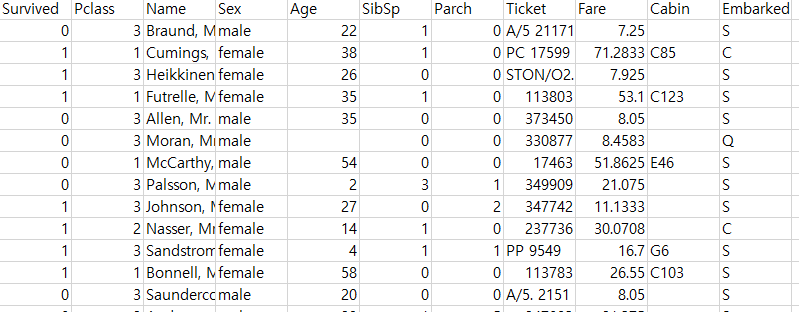

16.1 데이터 불러오기
# A tibble: 891 × 11
Survived Pclass Name Sex Age SibSp Parch Ticket Fare Cabin Embarked
<int> <int> <chr> <chr> <dbl> <int> <int> <chr> <dbl> <chr> <chr>
1 0 3 Braund, Mr. Owen Harris male 22 1 0 A/5 21171 7.25 "" S
2 1 1 Cumings, Mrs. John Bradley (Florence Briggs Thayer) female 38 1 0 PC 17599 71.3 "C85" C
3 1 3 Heikkinen, Miss. Laina female 26 0 0 STON/O2. 3101282 7.92 "" S
4 1 1 Futrelle, Mrs. Jacques Heath (Lily May Peel) female 35 1 0 113803 53.1 "C123" S
5 0 3 Allen, Mr. William Henry male 35 0 0 373450 8.05 "" S
6 0 3 Moran, Mr. James male NA 0 0 330877 8.46 "" Q
7 0 1 McCarthy, Mr. Timothy J male 54 0 0 17463 51.9 "E46" S
8 0 3 Palsson, Master. Gosta Leonard male 2 3 1 349909 21.1 "" S
9 1 3 Johnson, Mrs. Oscar W (Elisabeth Vilhelmina Berg) female 27 0 2 347742 11.1 "" S
10 1 2 Nasser, Mrs. Nicholas (Adele Achem) female 14 1 0 237736 30.1 "" C
# ℹ 881 more rows16.2 데이터 전처리 I
# 1. Convert to Factor
fac.col <- c("Pclass", "Sex")
titanic <- titanic %>%
data.frame() %>% # Data Frame 형태로 변환
mutate_at(fac.col, as.factor) # 범주형으로 변환
glimpse(titanic) # 데이터 구조 확인Rows: 891
Columns: 11
$ Survived <int> 0, 1, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 1, 0, 1, 1, 0, 1, 0, 0…
$ Pclass <fct> 3, 1, 3, 1, 3, 3, 1, 3, 3, 2, 3, 1, 3, 3, 3, 2, 3, 2, 3, 3, 2, 2, 3, 1, 3, 3, 3, 1, 3, 3, 1, 1, 3, 2, 1, 1, 3, 3, 3, 3, 3, 2, 3, 2, 3, 3, 3, 3, 3, 3, 3, 3, 1, 2, 1, 1, 2, 3, 2, 3, 3…
$ Name <chr> "Braund, Mr. Owen Harris", "Cumings, Mrs. John Bradley (Florence Briggs Thayer)", "Heikkinen, Miss. Laina", "Futrelle, Mrs. Jacques Heath (Lily May Peel)", "Allen, Mr. William Henry…
$ Sex <fct> male, female, female, female, male, male, male, male, female, female, female, female, male, male, female, female, male, male, female, female, male, male, female, male, female, femal…
$ Age <dbl> 22.0, 38.0, 26.0, 35.0, 35.0, NA, 54.0, 2.0, 27.0, 14.0, 4.0, 58.0, 20.0, 39.0, 14.0, 55.0, 2.0, NA, 31.0, NA, 35.0, 34.0, 15.0, 28.0, 8.0, 38.0, NA, 19.0, NA, NA, 40.0, NA, NA, 66.…
$ SibSp <int> 1, 1, 0, 1, 0, 0, 0, 3, 0, 1, 1, 0, 0, 1, 0, 0, 4, 0, 1, 0, 0, 0, 0, 0, 3, 1, 0, 3, 0, 0, 0, 1, 0, 0, 1, 1, 0, 0, 2, 1, 1, 1, 0, 1, 0, 0, 1, 0, 2, 1, 4, 0, 1, 1, 0, 0, 0, 0, 1, 5, 0…
$ Parch <int> 0, 0, 0, 0, 0, 0, 0, 1, 2, 0, 1, 0, 0, 5, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 1, 5, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 2, 2, 0…
$ Ticket <chr> "A/5 21171", "PC 17599", "STON/O2. 3101282", "113803", "373450", "330877", "17463", "349909", "347742", "237736", "PP 9549", "113783", "A/5. 2151", "347082", "350406", "248706", "38…
$ Fare <dbl> 7.2500, 71.2833, 7.9250, 53.1000, 8.0500, 8.4583, 51.8625, 21.0750, 11.1333, 30.0708, 16.7000, 26.5500, 8.0500, 31.2750, 7.8542, 16.0000, 29.1250, 13.0000, 18.0000, 7.2250, 26.0000,…
$ Cabin <chr> "", "C85", "", "C123", "", "", "E46", "", "", "", "G6", "C103", "", "", "", "", "", "", "", "", "", "D56", "", "A6", "", "", "", "C23 C25 C27", "", "", "", "B78", "", "", "", "", ""…
$ Embarked <chr> "S", "C", "S", "S", "S", "Q", "S", "S", "S", "C", "S", "S", "S", "S", "S", "S", "Q", "S", "S", "C", "S", "S", "Q", "S", "S", "S", "C", "S", "Q", "S", "C", "C", "Q", "S", "C", "S", "…# 2. Generate New Variable
titanic <- titanic %>%
mutate(FamSize = SibSp + Parch) # "FamSize = 형제 및 배우자 수 + 부모님 및 자녀 수"로 가족 수를 의미하는 새로운 변수
glimpse(titanic) # 데이터 구조 확인Rows: 891
Columns: 12
$ Survived <int> 0, 1, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 1, 0, 1, 1, 0, 1, 0, 0…
$ Pclass <fct> 3, 1, 3, 1, 3, 3, 1, 3, 3, 2, 3, 1, 3, 3, 3, 2, 3, 2, 3, 3, 2, 2, 3, 1, 3, 3, 3, 1, 3, 3, 1, 1, 3, 2, 1, 1, 3, 3, 3, 3, 3, 2, 3, 2, 3, 3, 3, 3, 3, 3, 3, 3, 1, 2, 1, 1, 2, 3, 2, 3, 3…
$ Name <chr> "Braund, Mr. Owen Harris", "Cumings, Mrs. John Bradley (Florence Briggs Thayer)", "Heikkinen, Miss. Laina", "Futrelle, Mrs. Jacques Heath (Lily May Peel)", "Allen, Mr. William Henry…
$ Sex <fct> male, female, female, female, male, male, male, male, female, female, female, female, male, male, female, female, male, male, female, female, male, male, female, male, female, femal…
$ Age <dbl> 22.0, 38.0, 26.0, 35.0, 35.0, NA, 54.0, 2.0, 27.0, 14.0, 4.0, 58.0, 20.0, 39.0, 14.0, 55.0, 2.0, NA, 31.0, NA, 35.0, 34.0, 15.0, 28.0, 8.0, 38.0, NA, 19.0, NA, NA, 40.0, NA, NA, 66.…
$ SibSp <int> 1, 1, 0, 1, 0, 0, 0, 3, 0, 1, 1, 0, 0, 1, 0, 0, 4, 0, 1, 0, 0, 0, 0, 0, 3, 1, 0, 3, 0, 0, 0, 1, 0, 0, 1, 1, 0, 0, 2, 1, 1, 1, 0, 1, 0, 0, 1, 0, 2, 1, 4, 0, 1, 1, 0, 0, 0, 0, 1, 5, 0…
$ Parch <int> 0, 0, 0, 0, 0, 0, 0, 1, 2, 0, 1, 0, 0, 5, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 1, 5, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 2, 2, 0…
$ Ticket <chr> "A/5 21171", "PC 17599", "STON/O2. 3101282", "113803", "373450", "330877", "17463", "349909", "347742", "237736", "PP 9549", "113783", "A/5. 2151", "347082", "350406", "248706", "38…
$ Fare <dbl> 7.2500, 71.2833, 7.9250, 53.1000, 8.0500, 8.4583, 51.8625, 21.0750, 11.1333, 30.0708, 16.7000, 26.5500, 8.0500, 31.2750, 7.8542, 16.0000, 29.1250, 13.0000, 18.0000, 7.2250, 26.0000,…
$ Cabin <chr> "", "C85", "", "C123", "", "", "E46", "", "", "", "G6", "C103", "", "", "", "", "", "", "", "", "", "D56", "", "A6", "", "", "", "C23 C25 C27", "", "", "", "B78", "", "", "", "", ""…
$ Embarked <chr> "S", "C", "S", "S", "S", "Q", "S", "S", "S", "C", "S", "S", "S", "S", "S", "S", "Q", "S", "S", "C", "S", "S", "Q", "S", "S", "S", "C", "S", "Q", "S", "C", "C", "Q", "S", "C", "S", "…
$ FamSize <int> 1, 1, 0, 1, 0, 0, 0, 4, 2, 1, 2, 0, 0, 6, 0, 0, 5, 0, 1, 0, 0, 0, 0, 0, 4, 6, 0, 5, 0, 0, 0, 1, 0, 0, 1, 1, 0, 0, 2, 1, 1, 1, 0, 3, 0, 0, 1, 0, 2, 1, 5, 0, 1, 1, 1, 0, 0, 0, 3, 7, 0…# 3. Select Variables used for Analysis
titanic1 <- titanic %>%
dplyr::select(Survived, Pclass, Sex, Age, Fare, FamSize) # 분석에 사용할 변수 선택
glimpse(titanic1) # 데이터 구조 확인Rows: 891
Columns: 6
$ Survived <int> 0, 1, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 1, 0, 1, 1, 0, 1, 0, 0…
$ Pclass <fct> 3, 1, 3, 1, 3, 3, 1, 3, 3, 2, 3, 1, 3, 3, 3, 2, 3, 2, 3, 3, 2, 2, 3, 1, 3, 3, 3, 1, 3, 3, 1, 1, 3, 2, 1, 1, 3, 3, 3, 3, 3, 2, 3, 2, 3, 3, 3, 3, 3, 3, 3, 3, 1, 2, 1, 1, 2, 3, 2, 3, 3…
$ Sex <fct> male, female, female, female, male, male, male, male, female, female, female, female, male, male, female, female, male, male, female, female, male, male, female, male, female, femal…
$ Age <dbl> 22.0, 38.0, 26.0, 35.0, 35.0, NA, 54.0, 2.0, 27.0, 14.0, 4.0, 58.0, 20.0, 39.0, 14.0, 55.0, 2.0, NA, 31.0, NA, 35.0, 34.0, 15.0, 28.0, 8.0, 38.0, NA, 19.0, NA, NA, 40.0, NA, NA, 66.…
$ Fare <dbl> 7.2500, 71.2833, 7.9250, 53.1000, 8.0500, 8.4583, 51.8625, 21.0750, 11.1333, 30.0708, 16.7000, 26.5500, 8.0500, 31.2750, 7.8542, 16.0000, 29.1250, 13.0000, 18.0000, 7.2250, 26.0000,…
$ FamSize <int> 1, 1, 0, 1, 0, 0, 0, 4, 2, 1, 2, 0, 0, 6, 0, 0, 5, 0, 1, 0, 0, 0, 0, 0, 4, 6, 0, 5, 0, 0, 0, 1, 0, 0, 1, 1, 0, 0, 2, 1, 1, 1, 0, 3, 0, 0, 1, 0, 2, 1, 5, 0, 1, 1, 1, 0, 0, 0, 3, 7, 0…16.3 데이터 탐색
ggpairs(titanic1,
aes(colour = as.factor(Survived))) + # Target의 범주에 따라 색깔을 다르게 표현
theme_bw()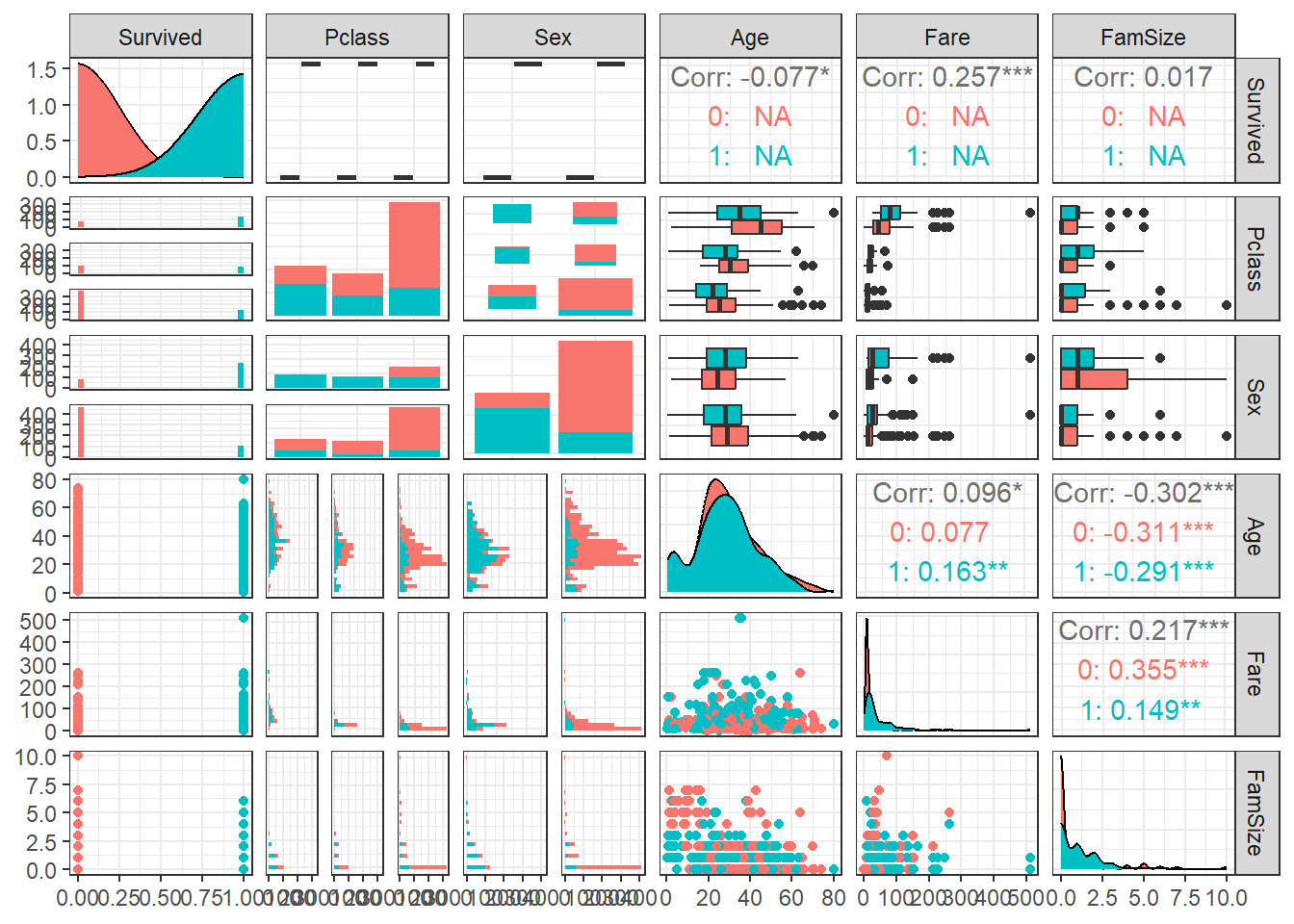
ggpairs(titanic1,
aes(colour = as.factor(Survived), alpha = 0.8)) + # Target의 범주에 따라 색깔을 다르게 표현
scale_colour_manual(values = c("#00798c", "#d1495b")) + # 특정 색깔 지정
scale_fill_manual(values = c("#00798c", "#d1495b")) + # 특정 색깔 지정
theme_bw()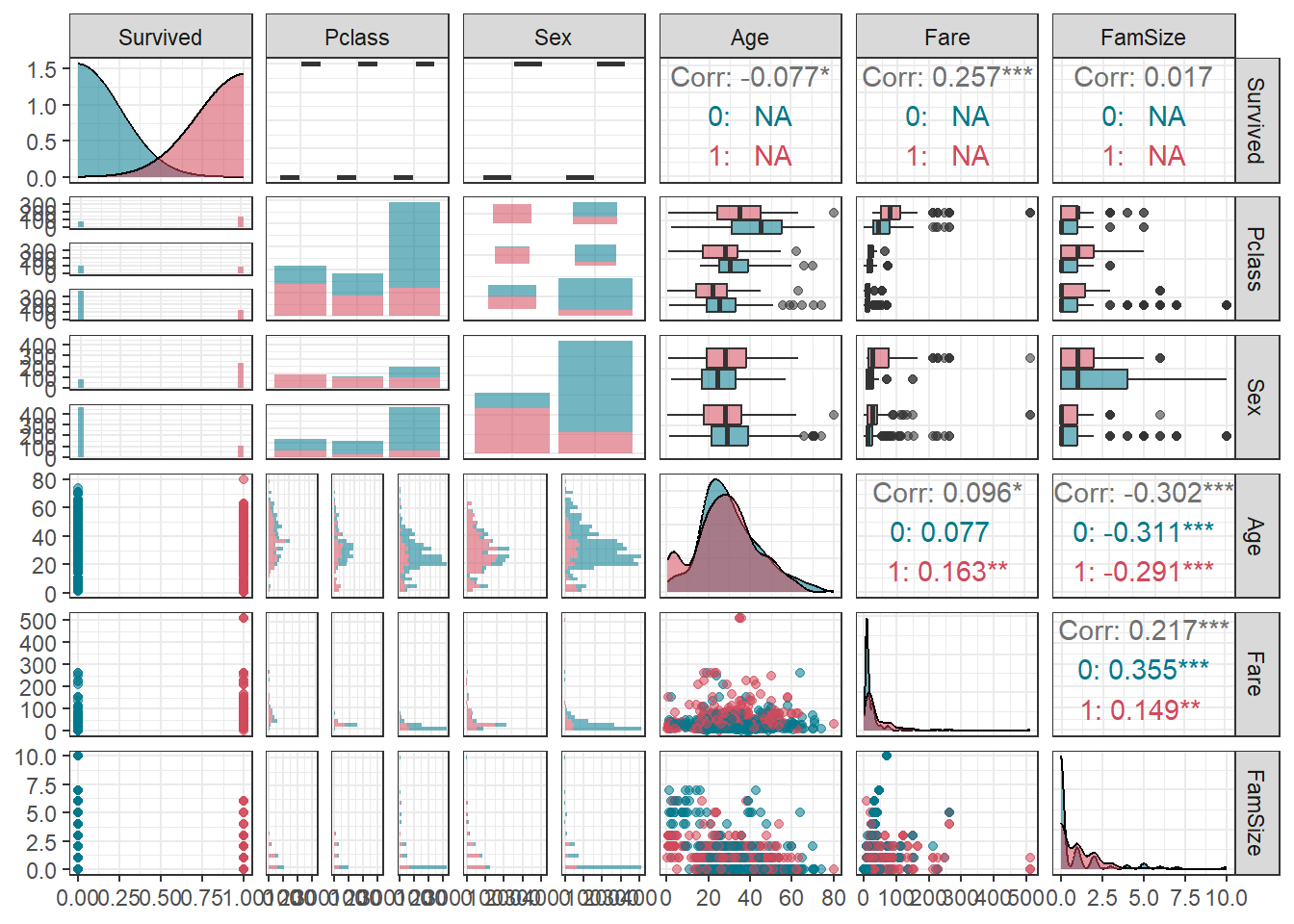
16.4 데이터 분할
# Partition (Training Dataset : Test Dataset = 7:3)
y <- titanic1$Survived # Target
set.seed(200)
ind <- createDataPartition(y, p = 0.7, list =T) # Index를 이용하여 7:3으로 분할
titanic.trd <- titanic1[ind$Resample1,] # Training Dataset
titanic.ted <- titanic1[-ind$Resample1,] # Test Dataset16.5 데이터 전처리 II
# Imputation
titanic.trd.Imp <- titanic.trd %>%
mutate(Age = replace_na(Age, mean(Age, na.rm = TRUE))) # 평균으로 결측값 대체
titanic.ted.Imp <- titanic.ted %>%
mutate(Age = replace_na(Age, mean(titanic.trd$Age, na.rm = TRUE))) # Training Dataset을 이용하여 결측값 대체
glimpse(titanic.trd.Imp) # 데이터 구조 확인Rows: 624
Columns: 6
$ Survived <int> 1, 1, 1, 0, 0, 1, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 1, 0, 0, 1, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 1, 1, 0, 1, 1, 0, 1, 0, 0, 0, 0, 0, 1, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1…
$ Pclass <fct> 1, 3, 1, 3, 1, 3, 2, 3, 3, 3, 3, 2, 3, 3, 3, 2, 2, 3, 1, 3, 1, 3, 3, 1, 1, 2, 1, 1, 3, 3, 3, 3, 3, 3, 3, 3, 3, 3, 1, 2, 1, 1, 2, 3, 2, 3, 3, 1, 3, 1, 3, 2, 3, 3, 2, 3, 3, 3, 3, 3, 2…
$ Sex <fct> female, female, female, male, male, female, female, female, male, male, female, female, male, female, female, male, male, female, male, female, male, female, male, male, female, mal…
$ Age <dbl> 38.00000, 26.00000, 35.00000, 35.00000, 54.00000, 27.00000, 14.00000, 4.00000, 20.00000, 39.00000, 14.00000, 55.00000, 2.00000, 31.00000, 29.92818, 35.00000, 34.00000, 15.00000, 28.…
$ Fare <dbl> 71.2833, 7.9250, 53.1000, 8.0500, 51.8625, 11.1333, 30.0708, 16.7000, 8.0500, 31.2750, 7.8542, 16.0000, 29.1250, 18.0000, 7.2250, 26.0000, 13.0000, 8.0292, 35.5000, 21.0750, 263.000…
$ FamSize <int> 1, 0, 1, 0, 0, 2, 1, 2, 0, 6, 0, 0, 5, 1, 0, 0, 0, 0, 0, 4, 5, 0, 0, 0, 1, 0, 1, 1, 0, 0, 2, 0, 0, 0, 1, 0, 5, 0, 1, 1, 1, 0, 0, 0, 3, 7, 0, 1, 5, 0, 2, 0, 0, 6, 0, 7, 0, 0, 0, 0, 0…glimpse(titanic.ted.Imp) # 데이터 구조 확인Rows: 267
Columns: 6
$ Survived <int> 0, 0, 0, 1, 1, 1, 0, 1, 1, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 1, 0, 1, 0…
$ Pclass <fct> 3, 3, 3, 1, 2, 3, 3, 3, 3, 3, 2, 2, 3, 3, 1, 3, 2, 3, 3, 3, 2, 3, 3, 1, 3, 3, 3, 1, 3, 3, 3, 3, 3, 2, 1, 3, 2, 3, 2, 3, 3, 2, 3, 3, 3, 3, 1, 3, 3, 3, 1, 3, 2, 3, 3, 1, 3, 3, 3, 3, 3…
$ Sex <fct> male, male, male, female, male, female, male, female, female, female, female, female, male, female, female, male, male, male, male, male, male, male, female, male, male, male, male,…
$ Age <dbl> 22.00000, 29.92818, 2.00000, 58.00000, 29.92818, 38.00000, 29.92818, 29.92818, 14.00000, 40.00000, 27.00000, 3.00000, 29.92818, 18.00000, 38.00000, 26.00000, 21.00000, 26.00000, 25.…
$ Fare <dbl> 7.2500, 8.4583, 21.0750, 26.5500, 13.0000, 31.3875, 7.2250, 7.7500, 11.2417, 9.4750, 21.0000, 41.5792, 21.6792, 17.8000, 80.0000, 8.6625, 73.5000, 14.4542, 7.6500, 7.8958, 29.0000, …
$ FamSize <int> 1, 0, 4, 0, 0, 6, 0, 0, 1, 1, 1, 3, 2, 1, 0, 2, 0, 1, 0, 0, 2, 0, 0, 0, 4, 0, 0, 1, 0, 0, 0, 1, 1, 0, 1, 0, 0, 0, 2, 0, 4, 0, 2, 1, 0, 0, 0, 0, 2, 4, 0, 0, 0, 6, 0, 0, 0, 0, 0, 0, 1…16.6 모형 훈련
Boosting은 다수의 약한 학습자(간단하면서 성능이 낮은 예측 모형)을 순차적으로 학습하는 앙상블 기법이다. Boosting의 특징은 이전 모형의 오차를 반영하여 다음 모형을 생성하며, 오차를 개선하는 방향으로 학습을 수행한다.
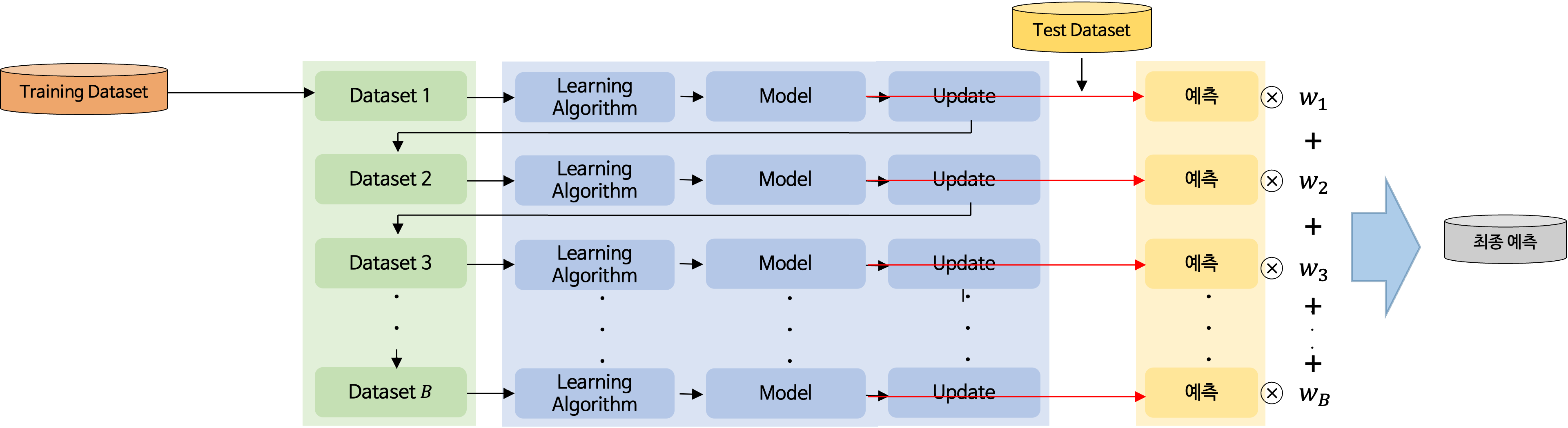
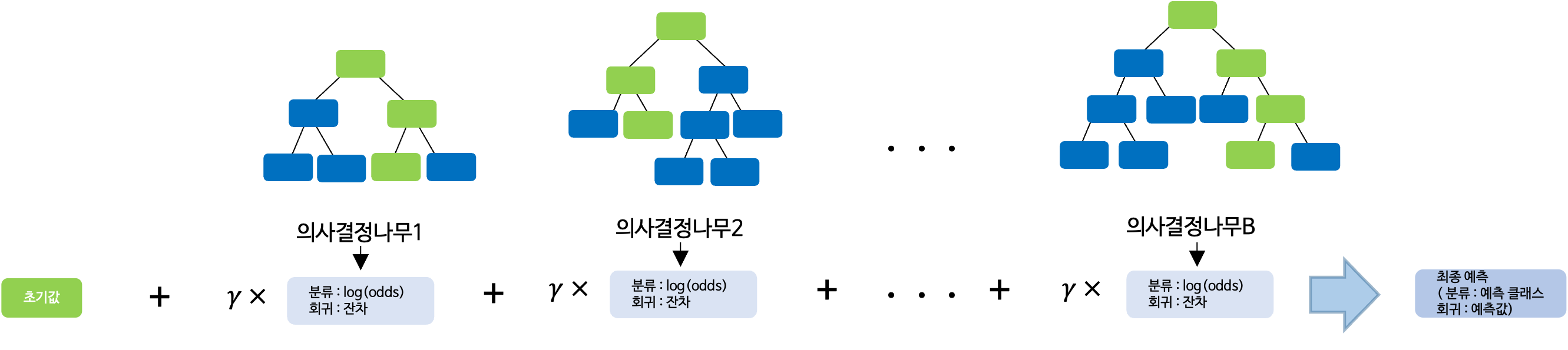
Gradient Boosting은 손실함수를 이용하여 손실함수가 작아지는 방향으로 예측값을 업데이트하며 이전 모형의 오차를 기반으로 다음 모형을 생성한다.

R에서 Gradient Boosting을 수행하기 위해 package "gbm"에서 제공하는 함수 gbm()를 이용할 수 있으며, 함수의 자세한 옵션은 여기를 참고한다.
gbm(formula, data, distribution, n.trees, interaction.depth, shrinkage, cv.folds, ...) formula: Target과 예측 변수의 관계를 표현하기 위한 함수로써 일반적으로Target ~ 예측 변수의 형태로 표현한다.data:formula에 포함하고 있는 변수들의 데이터셋(Data Frame)distribution: 손실함수- Classification :
"bernoulli"(이진 분류) - Regression :
"gaussian"(Squared Error)
- Classification :
n.trees: 생성하고자 하는 트리 개수interaction.depth: 트리의 최대 깊이shrinkage: 학습률cv.folds: \(k\)-Fold Cross Validation의 \(k\)(= Fold 수)- 값을 입력하면 Cross Validation를 통해
n.trees의 최적값을 찾을 수 있다.
- 값을 입력하면 Cross Validation를 통해
set.seed(100) # Seed 고정 -> 동일한 결과를 출력하기 위해
titanic.gbm <- gbm(Survived~.,
data = titanic.trd.Imp,
distribution = "bernoulli",
n.trees = 50,
interaction.depth = 30,
shrinkage = 0.1)Caution! 함수 gbm()을 사용하려면 이진 분류문제에서 Target은 "0" 또는 "1" 값을 가지는 수치형이어야 한다.
# 변수 중요도
summary.gbm(titanic.gbm, las = 2) 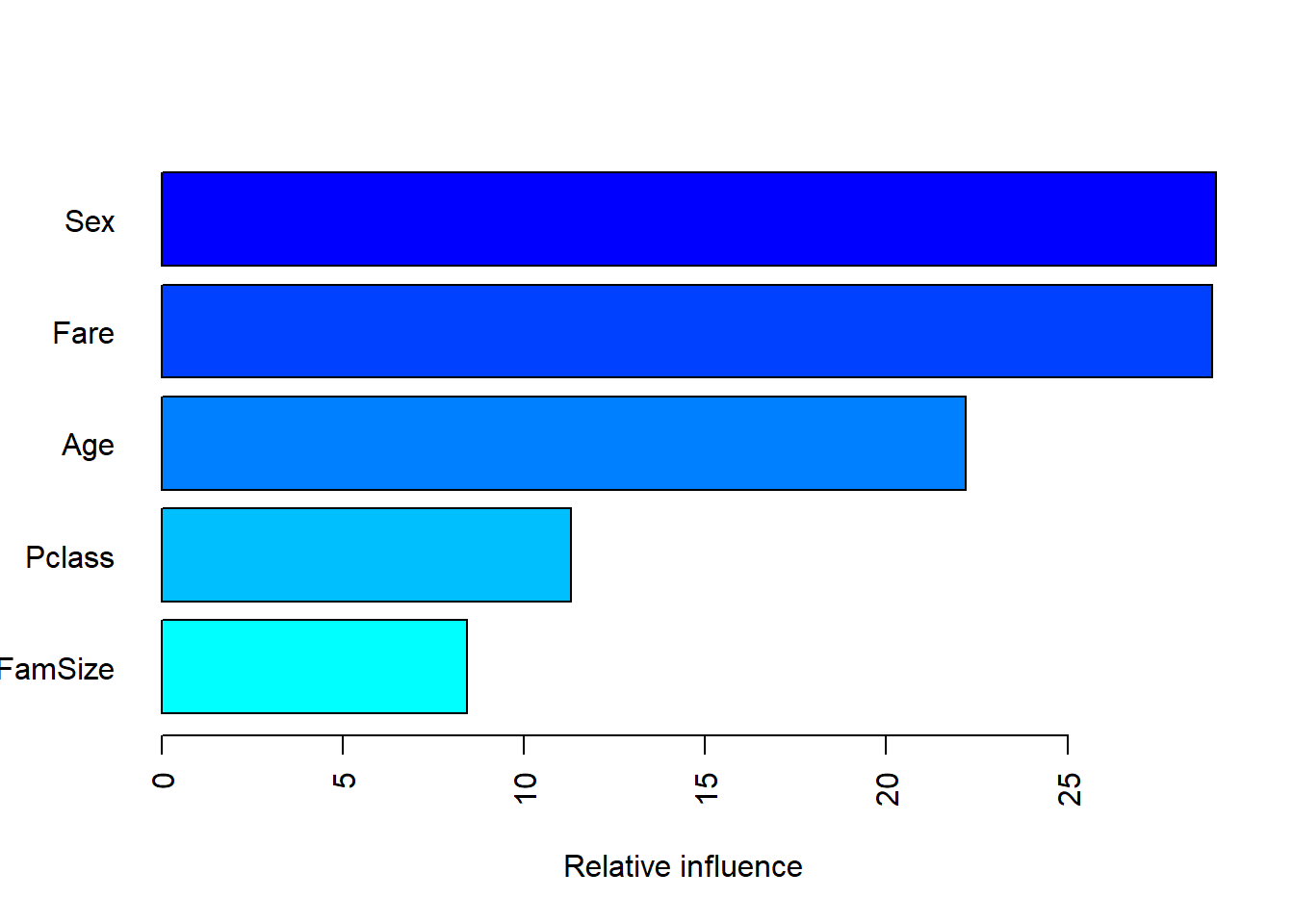
var rel.inf
Sex Sex 29.100291
Fare Fare 28.998586
Age Age 22.193140
Pclass Pclass 11.287106
FamSize FamSize 8.420878Result! 변수 Sex가 Target Survived을 분류하는 데 있어 중요하다.
16.7 모형 평가
Caution! 모형 평가를 위해 Test Dataset에 대한 예측 class/확률 이 필요하며, 함수 predict()를 이용하여 생성한다.
# 예측 확률 생성
test.gbm.prob <- predict(titanic.gbm,
newdata = titanic.ted.Imp[,-1],# Test Dataset including Only 예측 변수
type = "response") # "Survived = 1"에 대한 예측 확률 생성
test.gbm.prob %>%
as_tibble# A tibble: 267 × 1
value
<dbl>
1 0.113
2 0.0571
3 0.412
4 0.936
5 0.249
6 0.0670
7 0.0433
8 0.655
9 0.801
10 0.215
# ℹ 257 more rows16.7.1 ConfusionMatrix
# 예측 class 생성
cv <- 0.5 # Cutoff Value
test.gbm.class <- as.factor(ifelse(test.gbm.prob > cv, "1", "0")) # 예측 확률 > cv이면 "Survived = 1" 아니면 "Survived = 0"
test.gbm.class %>%
as_tibble# A tibble: 267 × 1
value
<fct>
1 0
2 0
3 0
4 1
5 0
6 0
7 0
8 1
9 1
10 0
# ℹ 257 more rowsCM <- caret::confusionMatrix(test.gbm.class, as.factor(titanic.ted.Imp$Survived),
positive = "1") # confusionMatrix(예측 class, 실제 class, positive = "관심 class")
CMConfusion Matrix and Statistics
Reference
Prediction 0 1
0 156 26
1 12 73
Accuracy : 0.8577
95% CI : (0.8099, 0.8973)
No Information Rate : 0.6292
P-Value [Acc > NIR] : < 2e-16
Kappa : 0.6859
Mcnemar's Test P-Value : 0.03496
Sensitivity : 0.7374
Specificity : 0.9286
Pos Pred Value : 0.8588
Neg Pred Value : 0.8571
Prevalence : 0.3708
Detection Rate : 0.2734
Detection Prevalence : 0.3184
Balanced Accuracy : 0.8330
'Positive' Class : 1
16.7.2 ROC 곡선
ac <- titanic.ted.Imp$Survived # Test Dataset의 실제 class
pp <- as.numeric(test.gbm.prob) # 예측 확률을 수치형으로 변환16.7.2.1 Package “pROC”
pacman::p_load("pROC")
gbm.roc <- roc(ac, pp, plot = T, col = "gray") # roc(실제 class, 예측 확률)
auc <- round(auc(gbm.roc), 3)
legend("bottomright", legend = auc, bty = "n")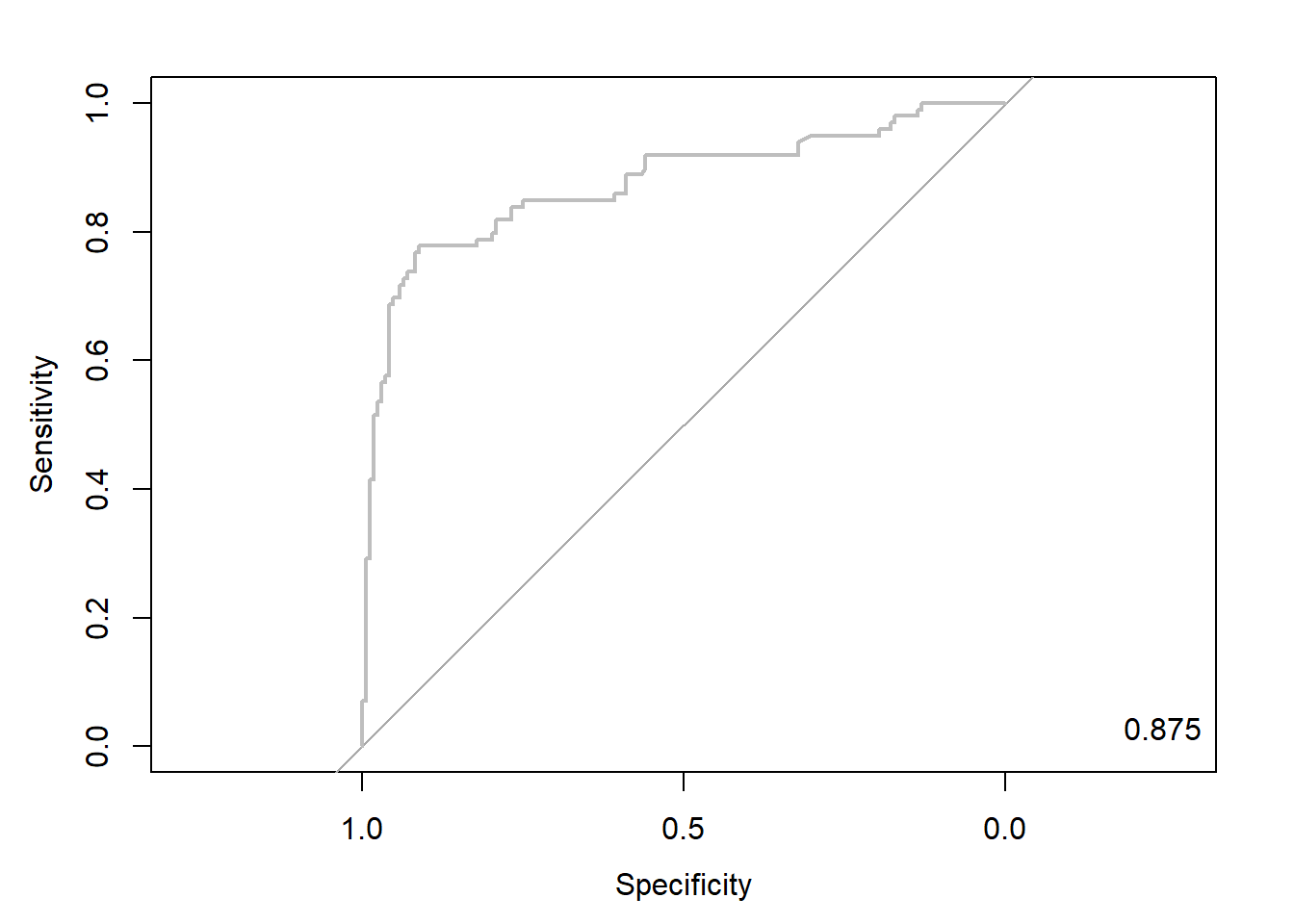
Caution! Package "pROC"를 통해 출력한 ROC 곡선은 다양한 함수를 이용해서 그래프를 수정할 수 있다.
# 함수 plot.roc() 이용
plot.roc(gbm.roc,
col="gray", # Line Color
print.auc = TRUE, # AUC 출력 여부
print.auc.col = "red", # AUC 글씨 색깔
print.thres = TRUE, # Cutoff Value 출력 여부
print.thres.pch = 19, # Cutoff Value를 표시하는 도형 모양
print.thres.col = "red", # Cutoff Value를 표시하는 도형의 색깔
auc.polygon = TRUE, # 곡선 아래 면적에 대한 여부
auc.polygon.col = "gray90") # 곡선 아래 면적의 색깔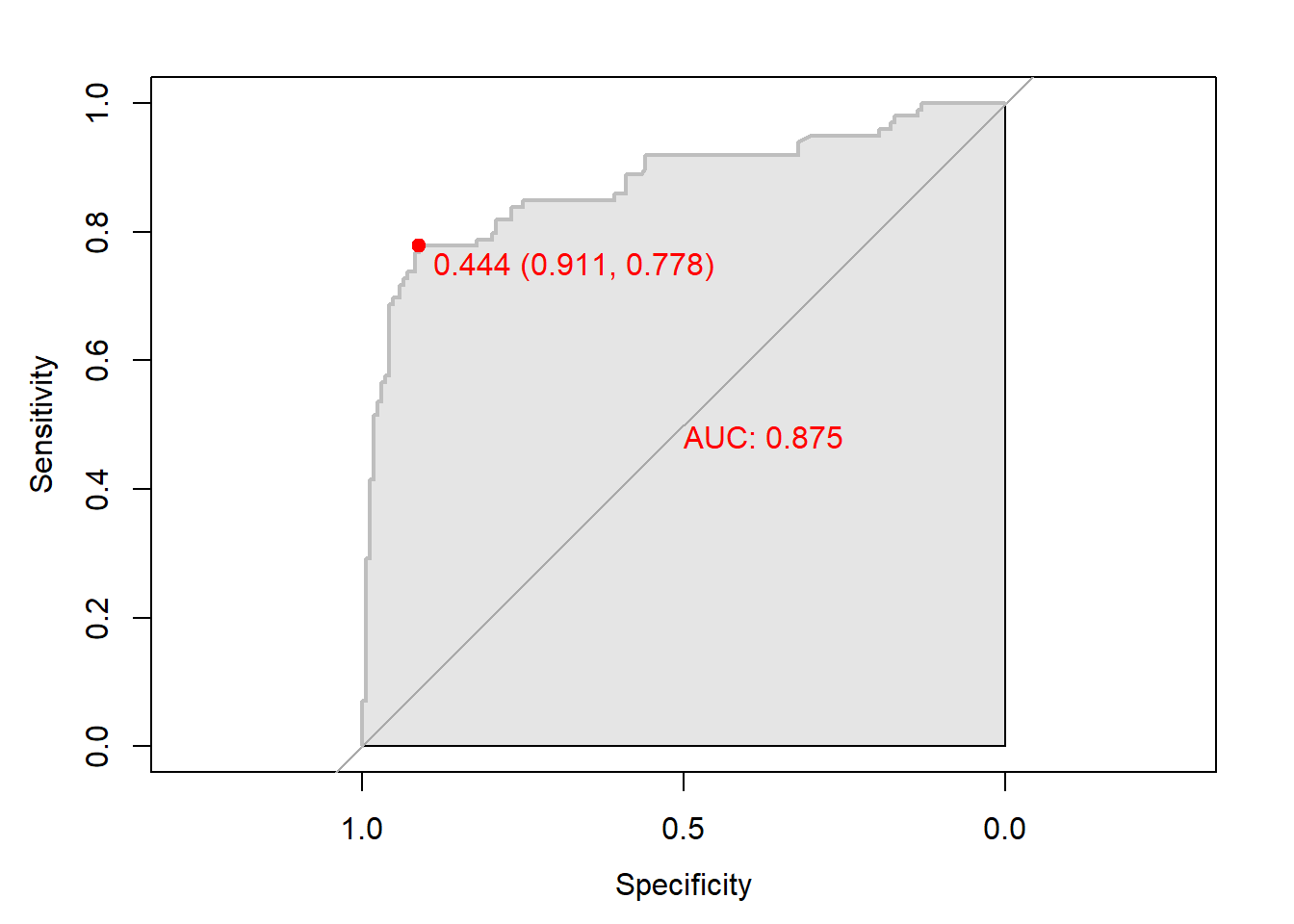
# 함수 ggroc() 이용
ggroc(gbm.roc) +
annotate(geom = "text", x = 0.9, y = 1.0,
label = paste("AUC = ", auc),
size = 5,
color="red") +
theme_bw()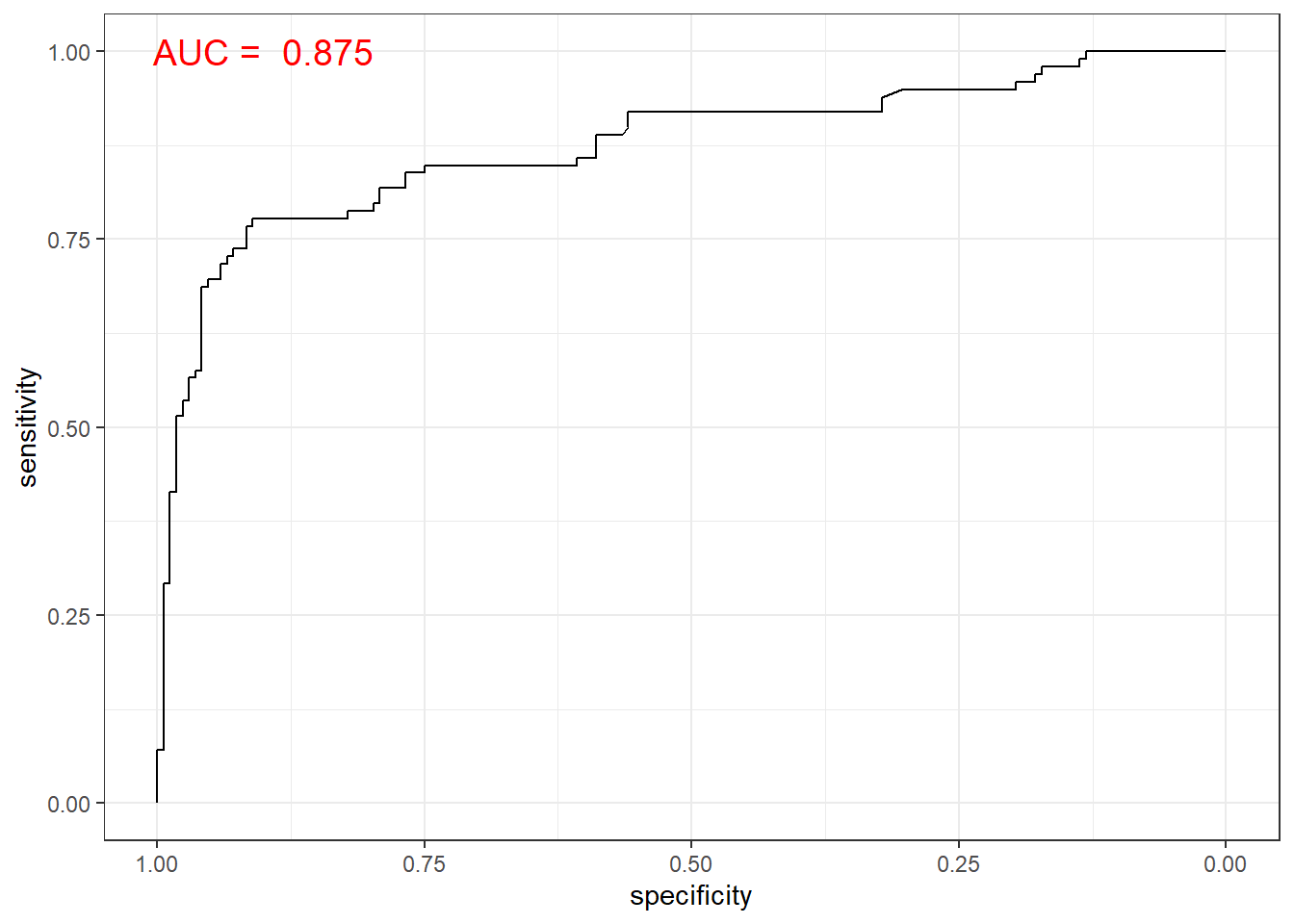
16.7.2.2 Package “Epi”
pacman::p_load("Epi")
# install_version("etm", version = "1.1", repos = "http://cran.us.r-project.org")
ROC(pp, ac, plot = "ROC") # ROC(예측 확률, 실제 class) 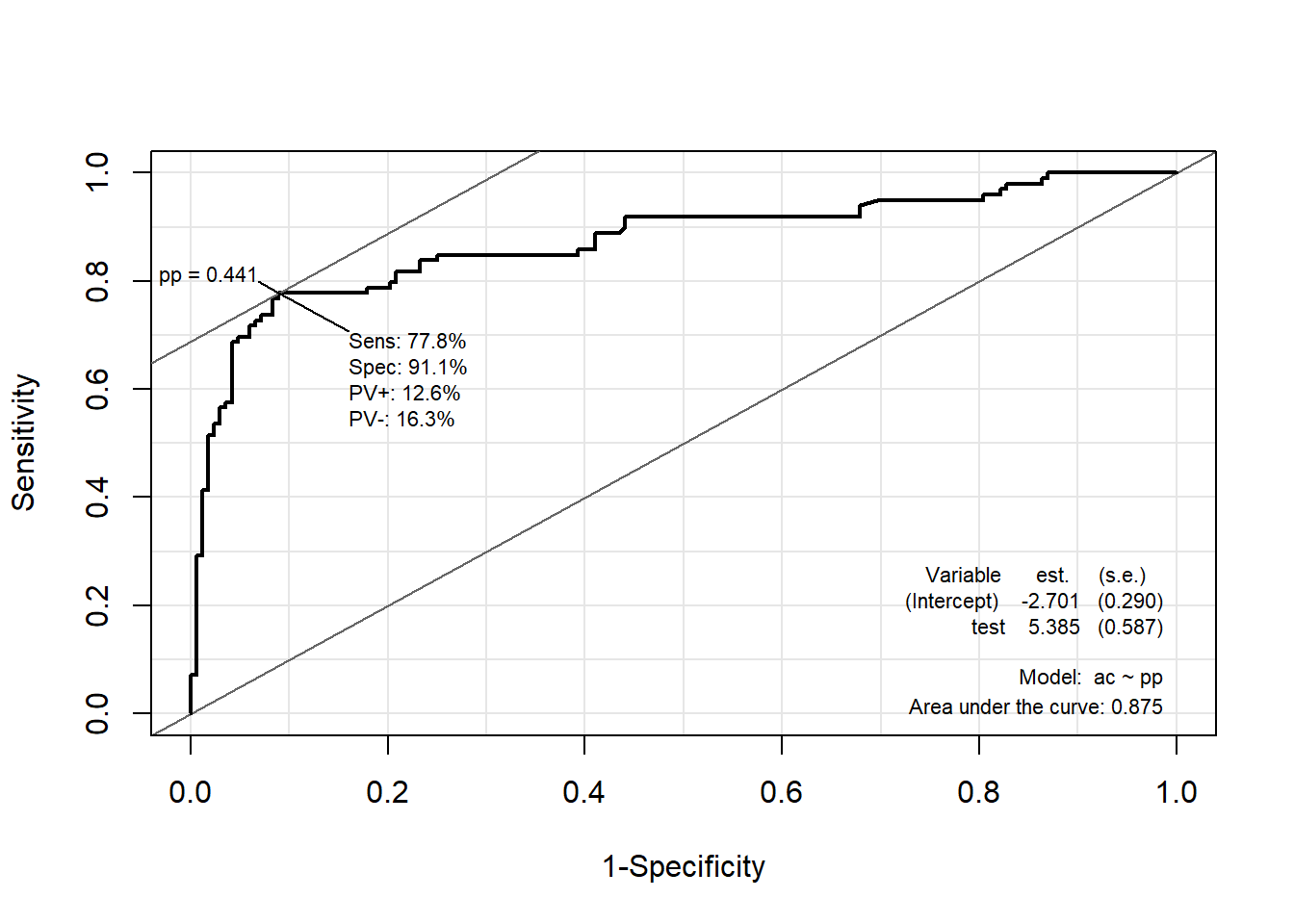
16.7.2.3 Package “ROCR”
pacman::p_load("ROCR")
gbm.pred <- prediction(pp, ac) # prediction(예측 확률, 실제 class)
gbm.perf <- performance(gbm.pred, "tpr", "fpr") # performance(, "민감도", "1-특이도")
plot(gbm.perf, col = "gray") # ROC Curve
perf.auc <- performance(gbm.pred, "auc") # AUC
auc <- attributes(perf.auc)$y.values
legend("bottomright", legend = auc, bty = "n")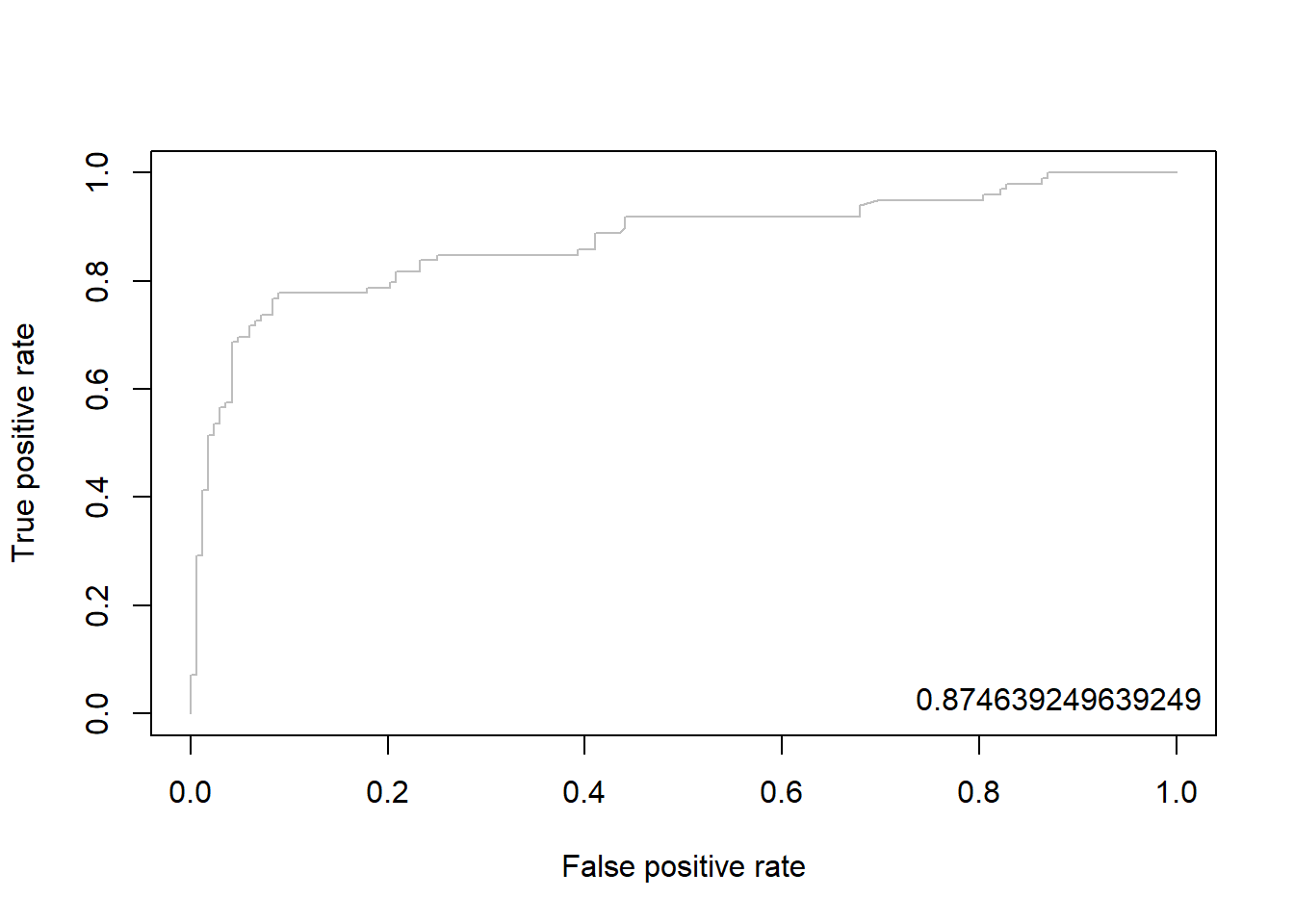
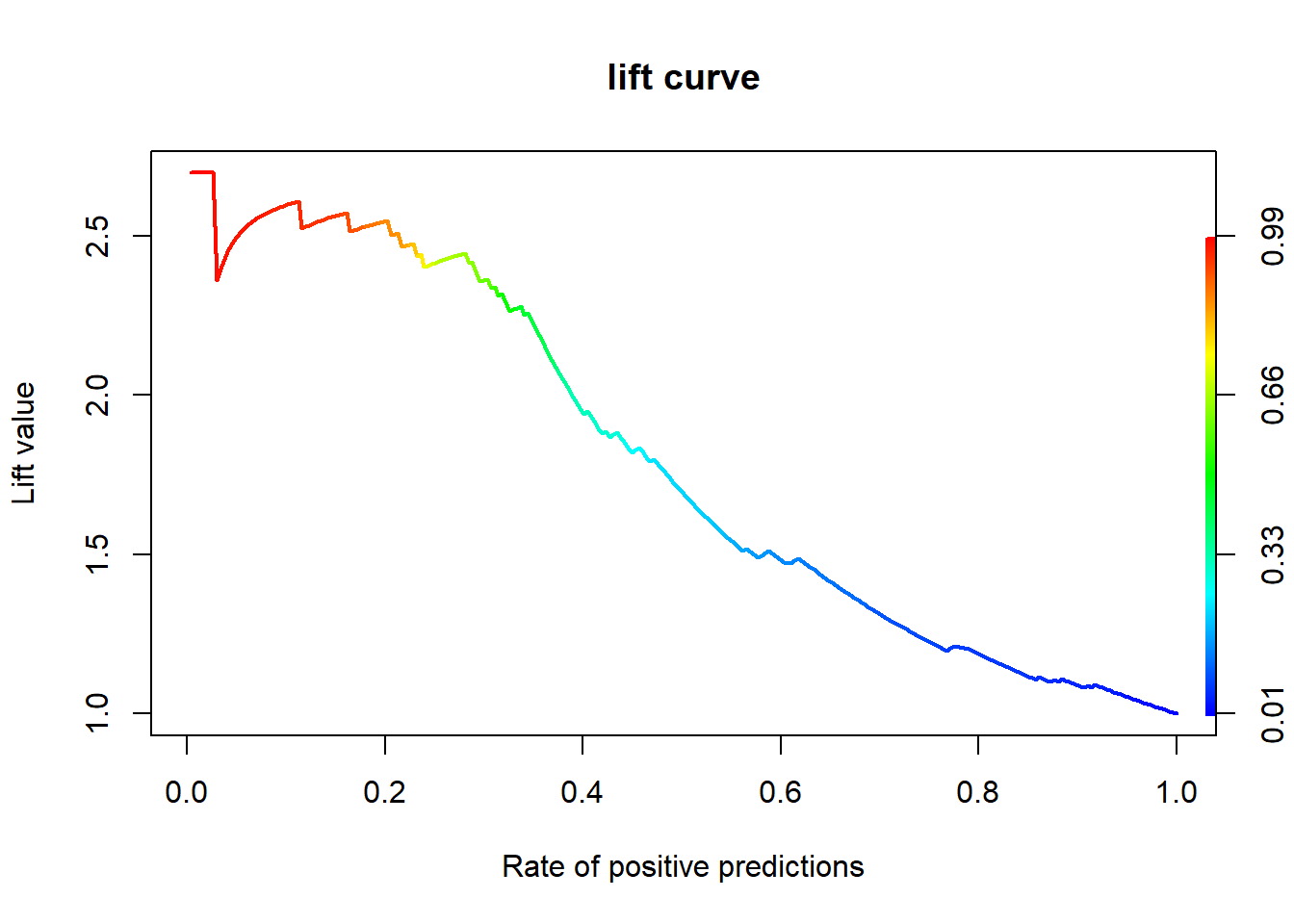
16.7.3 향상 차트
16.7.3.1 Package “ROCR”
gbm.perf <- performance(gbm.pred, "lift", "rpp") # Lift Chart
plot(gbm.perf, main = "lift curve",
colorize = T, # Coloring according to cutoff
lwd = 2)